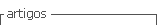Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Iatreia
versão impressa ISSN 0121-0793
Iatreia v.21 n.4 Medellín out./dez. 2008
ARTÍCULO DE INVESTIGACIÓN
Estudio de las aneuploidías del cromosoma 17 y la deleción del gen TP53 en neoplasias hematológicas, por la técnica del FISH–bicolor
Study of chromosome 17 aneuploidy and of TP53 gene deletion in patients with hematological neoplasias by dualcolor fluorescence in situ hybridization (FISH)
Juan Carlos Herrera Patiño1; Gloria Cecilia Ramírez Gaviria1; Carlos Mario Muñetón Peña2
1.Bacteriólogo, Unidad de Genética Médica, Departamento de Educación Médica, Facultad de Medicina, Universidad de Antioquia, Medellín, Colombia.
2. Docente, Unidad de Genética Médica, Departamento de Educación Médica, Facultad de Medicina, Universidad de Antioquia, Medellín, Colombia.
Dirección de contacto: cmuneton@quimbaya.udea.edu.co
Resumen
Las neoplasias hematológicas se caracterizan por presentar una amplia diversidad de alteraciones genéticas. Se analizaron 15 muestras de pacientes con diferentes tipos de neoplasias hematológicas mediante la técnica FISH, para detectar aneuploidías del cromosoma 17 y la deleción del gen TP53. En 11 de ellas se hicieron análisis cromosómicos por citogenética convencional; 6 de las 11 tenían cariotipo anormal (54,5%): se detectaron 3 translocaciones y 3 mosaicismos. El análisis de las 15 muestras mediante la técnica FISH mostró un 26,7% de aneuploidía del cromosoma 17 y un 33,3% con deleción del gen TP53. De los 6 casos con cariotipo anormal, en 2 se detectaron alteraciones por FISH. En 5 casos se detectaron con esta técnica alteraciones cromosómicas no observadas por citogenética convencional. Solo en 3 (20%) de las 15 muestras analizadas el análisis cromosómico resultó normal por citogenética convencional y FISH. En este trabajo se corrobora que la aneuploidía del cromosoma 17 y la deleción del gen TP53 tienen una baja frecuencia en las neoplasias hematológicas. Sin embargo, el valor pronóstico de estas alteraciones genéticas no está bien definido.
Palabras clave: Aneuploidía, Deleción, Gen TP53, Hibridación in situ por fluorescencia (HISF), Inestabilidad genética, Neoplasias hematológicas
Summary
Hematological neoplasias are characterized by a wide spectrum of genetic alterations. We analyzed 15 specimens from patients with various types of hematological malignancies by means of the FISH technique in order to detect aneuploidy of chromosome 17 and deletion of TP53 gene. In 11 of them chromosomal analyses were also carried out using conventional cytogenetic techniques; in 6 of these 11 specimens (54.5%) abnormal karyotypes were detected, namely: 3 translocations and 3 mosaicisms. FISH results revealed that in 26.7% of the 15 specimens there was chromosome 17 aneuploidy, and that 33.3% had TP53 deletion. Out of the 6 cases with abnormal karyotypes, further alterations were detected in two by FISH. In 5 cases chromosomal abnormalities were detected by FISH but not by the conventional cytogenetic procedures. Only in 3 (20%) out of the 15 specimens the results of chromosomal analyses were normal by both the conventional cytogenetics and FISH. These results corroborate the low frequency of chromosome 17 aneuploidy and of TP53 gene deletion in hematological neoplasias. However, the prognostic value of these genetic alterations is still not well defined.
Key words: Aneuploidy, Deletion, FISH, Genetic instability, Hematological neoplasias, TP53 gene
INTRODUCCIÓN
Las neoplasias hematológicas tienen origen clonal y se caracterizan por presentar una gran heterogeneidad genética; hay una amplia variación geográfica de las alteraciones genéticas relacionadas con ellas, posiblemente por factores ambientales o por la composición genética de las poblaciones.1 Muchos mecanismos genéticos se relacionan con el origen de las neoplasias hematológicas, entre ellos: mutaciones puntuales, pérdida de heterocigocidad (LOH, por su sigla en inglés), amplificación génica, metilación de genes y alteraciones cromosómicas numéricas (aneuploidías) y estructurales (principalmente deleciones, inversiones y translocaciones).1,2 Estas alteraciones genéticas y epigenéticas conducen a la activación de protooncogenes o a la inactivación de genes supresores de tumores, que por estos mecanismos promueven la inestabilidad genómica. Estos dos grupos de genes son esenciales en el control de la proliferación y el ciclo celulares.
El análisis citogenético de las neoplasias hematológicas es importante para el diagnóstico y el pronóstico, así como para el conocimiento de las bases genéticas de su patogénesis.3 Específicamente en leucemias y linfomas, la citogenética convencional tiene un papel significativo en el estudio de la genética del cáncer, puesto que ha permitido identificar un gran número de marcadores cromosómicos que se repiten en casi todos los pacientes afectados por cánceres hematológicos, como la translocación t(9;22) (q34;q11) o el cromosoma Filadelfia (Ph1), una de las alteraciones citogenéticas de gran importancia para el diagnóstico, tratamiento y seguimiento de la leucemia mieloide crónica.4
Con el desarrollo de la citogenética molecular, la hibridación in situ por fluorescencia (FISH, por su sigla en inglés) revolucionó los análisis cromosómicos en la genética del cáncer. Esta prueba se ha convertido en uno de los avances más importantes en el diagnóstico citogenético de neoplasias hematológicas, especialmente porque permite analizar células en interfase (I–FISH).5 Su sensibilidad y especificidad son altas por lo que con ella se logra identificar un gran número de reordenamientos cromosómicos en regiones que contienen genes implicados en el desarrollo de neoplasias hematológicas, que no se detectaban con la citogenética convencional. Además, la técnica FISH tiene otras ventajas como la de hacer el análisis cromosómico en un tiempo corto y analizar un mayor número de células por caso; esto la diferencia de la citogenética convencional cuyas desventajas son: el bajo índice mitótico de los cultivos celulares, la morfología pobre de los cromosomas y un bandeo cromosómico deficiente, lo que dificulta la detección de ciertas alteraciones cromosómicas estructurales y numéricas.5,6 En la actualidad están disponibles numerosas sondas comerciales para detectar alteraciones cromosómicas recurrentes y que permiten también evaluar cambios en el número de copias de genes específicos en neoplasias hematológicas y tumores sólidos.7 Por lo anterior, se ha implementado la técnica FISH como de rutina en los laboratorios de citogenética, para el diagnóstico molecular de alteraciones cromosómicas y génicas en diversas neoplasias.
De otro lado, numerosos estudios citogenéticos llevados a cabo en neoplasias hematológicas con las técnicas convencionales y moleculares han hallado una gran variedad de alteraciones cromosómicas y con ellos se ha demostrado que la inestabilidad cromosómica es común en leucemias y linfomas.3,4,8,9
Entre las anormalidades cromosómicas más frecuentes en las neoplasias hematológicas están las deleciones en las regiones cromosómicas 13q14, 11q22–q23, 17p13 y 5q–; aneuploidías de los cromosomas 7, 8, 11 y 17 y translocaciones como la t(9;22), t(8;21).1,5,8 Cabe mencionar que en estas regiones cromosómicas se localizan protooncogenes y genes supresores de tumores, que son importantes para mantener la estabilidad genómica de las células.
Dichas alteraciones cromosómicas tienen implicaciones importantes para el pronóstico, puesto que en muchos casos se asocian con las manifestaciones clínicas y patológicas de la enfermedad, un mal pronóstico, períodos cortos de supervivencia y fallas en la respuesta a determinados tratamientos.8
Específicamente la deleción 17p13 es una alteración cromosómica común en las neoplasias hematológicas cuya frecuencia parece variar con el avance de la enfermedad.1,8,10 Así, mientras que algunos autores la han informado en porcentajes bajos,11 otros la han hallado en porcentajes altos en los estados avanzados de la enfermedad. Debe tenerse en cuenta que en el locus 17p13.1 se localiza el gen supresor de tumores TP53, que codifica para una fosfoproteína nuclear de 53 kd (proteína p53), que actúa como factor de transcripción.12 Este gen interviene en muchas funciones celulares, especialmente en el control del ciclo, la apoptosis, la respuesta al daño y la reparación del ADN, por lo que también se lo conoce como 'gen guardián del genoma humano'.13 El gen TP53 se encuentra alterado en más del 50% de las neoplasias estudiadas, con mayor frecuencia en los tumores sólidos que en las leucemias y linfomas.12,13 La deleción 17p13.1 y las mutaciones en el gen TP53 se asocian con un mal pronóstico y con los estadios avanzados de malignidad en diversas neoplasias.14
El objetivo del presente trabajo fue evaluar las aneuploidías del cromosoma 17 y la deleción del gen TP53 (locus 17p13.1) en 15 pacientes con neoplasias hematológicas, empleando la técnica FISH bicolor.
MATERIALES Y MÉTODOS
Pacientes
Se analizaron muestras de médula ósea o sangre periférica de 15 pacientes con neoplasias hematológicas. Once fueron mujeres (edad promedio: 39 años; rango: 6 a 63 años) y cuatro, hombres (edad promedio: 17 años; rango: 14 a 19 años). Previo consentimiento informado por escrito, se obtuvieron las muestras mediante aspirado de médula ósea o flebotomía, en los servicios de hematooncología de la Facultad de Medicina de la Universidad de Antioquia, el Hospital Universitario San Vicente de Paúl y el Hospital Pablo Tobón Uribe, instituciones, todas ellas, de la ciudad de Medellín. Las muestras se remitieron al Laboratorio de Genética Médica, de la Facultad de Medicina de la Universidad de Antioquia. Los pacientes no habían recibido previamente quimioterapia ni radioterapia. El estudio fue aprobado por el Comité de Bioética de la Universidad de Antioquia.
Estudio citogenético
Las muestras de médula ósea y sangre periférica se recolectaron en jeringas heparinizadas de 10 mL. Se establecieron cultivos celulares en el medio HAMF12/DMD (Sigma–Aldrich, St. Louis, MO), suplementado con 10% de suero bovino fetal (GibcoBRL, Grand Island, NY) y se incubaron a 37 ºC por 24 horas. Luego se adicionaron 100 μl de Colcemid®, a 37 ºC por 10 minutos, se centrifugó a 1.200 rpm por 10 minutos y se descartó el sobrenadante. Inmediatamente después se adicionó una solución hipotónica (KCl al 0,075 M; J. T. Baker, Phillipsburgh, NJ) y se incubó a 37 ºC por 15 minutos; después se centrifugó en las mismas condiciones ya descritas y se descartó el sobrenadante; posteriormente se hizo una prefijación con 10 mL del fijador metanol/ácido acético 3:1 a temperatura ambiente por 15 minutos, se centrifugó durante 10 minutos a 1.200 rpm y se descartó el sobrenadante, y seguidamente se hicieron tres lavados con el mismo fijador. Finalmente, se depositaron gotas de la suspensión celular en los portaobjetos. En todos estos se realizó bandeo R. Para cada caso se analizaron al menos 20 metafases; los cariotipos se organizaron según la nomenclatura del ISCN (del inglés: International Standing Committee on Human Cytogenetic Nomenclature) 2005. Las placas para el FISH se almacenaron a –20 ºC hasta el momento del proceso.
Técnica FISH bicolor
La técnica FISH bicolor se hizo con sondas comerciales marcadas directamente con diversos fluorocromos: para detectar aneuploidías del cromosoma 17 se utilizó una sonda centromérica (CEP 17, señal verde, Vysis–Abbott); para detectar la deleción del gen TP53 se utilizó una sonda de locus específico para este gen (LSI 17p13.1, señal naranja,Vysis–Abbott). Para las condiciones de pretratamiento de las placas, hibridación y análisis se siguieron las instrucciones y recomendaciones del fabricante (Vysis–Abbott). Brevemente descritas, los extendidos se desnaturalizaron en formamida al 70% a 73 ºC por 5 minutos; luego se deshidrataron en una serie de etanoles fríos al 70%, 85% y 100%. A cada placa se le adicionó una mezcla de10 μl de cada una de las sondas CEP 17 y LSI 17p13.1 en el área de hibridación seleccionada, sobre la cual se puso un cubreobjetos de 22 x 22 mm. La hibridación se hizo en una cámara húmeda incubada a 37 ºC por 16 horas. Después de ella se lavó dos veces. Finalmente, las placas se colorearon con DAPI (sigla en inglés de 4,6–diamino–2–fenilindol) de Vysis–Abbott. Las señales de hibridación se analizaron en un microscopio de epifluorescencia (Axyoskop 2 plus Zeiss, Alemania) dotado con un filtro triple simultáneo (DAPI/ORANGE/GREEN) y las imágenes se captaron con una cámara CCD AxioCam MRc (Zeiss). En cada caso se analizaron 100 células, excluyendo del análisis los núcleos fusionados o con señales difusas. Como control se incluyeron muestras de sangre periférica de 5 individuos sanos. En los controles el patrón de hibridación fue de dos señales verdes (cromosoma 17) y dos de color naranja (locus 17p13.1 del gen TP53), para un total de cuatro señales de hibridación.
Con base en análisis previos hechos con las muestras de control (datos no presentados), se establecieron los siguientes puntos de corte para el análisis del FISHbicolor: aneuploidía cuando más del 9% de las células presentaban menos o más de 2 señales de hibridación para el centrómero del cromosoma 17; es decir, que una muestra es normal (disómica) si tiene un porcentaje del 91% o más de los núcleos con dos señales para el centrómero del cromosoma 17; para la nulisomía, la trisomía y la monosomía los puntos de corte fueron del 1%, 4% y 7%, respectivamente. La tetrasomía no se observó en los núcleos normales, por lo que se definió arbitrariamente el 1% como punto de corte. En todos los casos los puntos de corte se calcularon con dos desviaciones estándar. Cuando se detectaron subpoblaciones de células con clones heterogéneos (monosómicos/disómicos/ o trisómicos/tetrasómicos), se estableció que el resultado final se informaría con base en el clon mayor, es decir, en el que presentara el mayor porcentaje de células anormales.
La deleción en el locus 17p.13.1 del gen TP53 se determinó dividiendo el total de señales de hibridación del gen TP53 por el total de señales del centrómero del cromosoma 17 (Total de señales LSI TP53 /Total de señales CEP 17). Se definió que una muestra tenía deleción del gen TP53 cuando se obtenía un valor de 0,95 o menor.
RESULTADOS
En la tabla n.º 1 se presenta la información general de cada uno de los pacientes estudiados: edad, sexo, tipo de neoplasia hematológica y los resultados obtenidos por citogenética convencional y FISH.
El estudio citomorfológico de los aspirados medulares y la sangre periférica mostró cinco casos de leucemia mieloide aguda (33,3%), tres de leucemia mieloide crónica (20%), dos leucemias linfoides agudas (13,3%), dos leucemias eosinofílicas crónicas (13,3%), un caso de leucemia linfoide crónica, uno de síndrome mielodisplásico y uno de trombocitemia esencial (Tabla n.º 1).
En 13 de las 15 muestras se establecieron cultivos celulares para el estudio cromosómico convencional por citogenética, mientras que los otros dos casos se estudiaron por la técnica FISH para evaluar la presencia del cromosoma Filadelfia. En dos muestras no se obtuvieron metafases para el estudio citogenético. De los 11 casos restantes se encontró que 5 (45,5%) presentaban un cariotipo normal (46,XX o 46,XY), mientras que 6 (54,5%) lo tenían normal (Tabla n.º 1). De estos últimos, 3 presentaban translocaciones (casos 02, 03, 14): dos de ellos tenían 46,XX, t(9;22) y uno, 45,X, t(8;21), mientras que los otros 3 (casos 01, 12, 15) tenían mosaicismos, o sea, alteraciones numéricas compuestas por dos líneas celulares diferentes, una normal y otra con hipodiploidía (Tabla n.º 1).
En cuanto a los resultados obtenidos por la técnica FISH, se encontró que 4 de las 15 muestras (26,7%) tenían aneuploidías del cromosoma 17 y 11 (73,3%) eran normales para el número de copias de este cromosoma (Tabla n.º 1). De los 4 casos con aneuploidías, 3 tenían una monosomía (75%) (casos 03, 05 y 13) y el otro presentaba tetrasomía del cromosoma 17 (caso 04), el cual se destacó por presentar un alto porcentaje (14%) de células con cuatro señales para este cromosoma (Figura n.º 1). En ninguno de los casos se detectaron núcleos con pérdida de las dos copias del cromosoma 17 (nulisomía).
La deleción en el locus 17p.13.1 del gen TP53 se observó en 5 de las 15 muestras (33,3%) analizadas por la técnica FISH (casos 01, 03, 04, 06 y 11); las 10 restantes (66,7%) eran normales para el número de copias del gen TP53 (Tabla n.º 1) (Figura n.º 1). De acuerdo con estos resultados, se definió que la deleción monoalélica fue la pérdida común en todos los casos. El caso 04 presentó un alto porcentaje (10%) de núcleos con cuatro copias del gen TP53, similar a lo observado para la aneuploidía del cromosoma 17.
En la mayoría de los casos analizados con la técnica FISH–bicolor se detectó la presencia simultánea de subpoblaciones de núcleos con clones monosómicos, disómicos y trisómicos y en menor proporción tetrasómicos (Figura n.º 1).
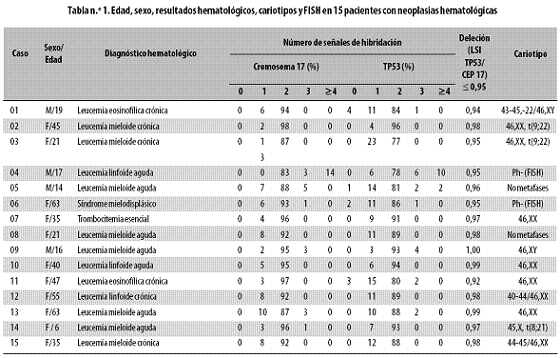
Comparando los resultados obtenidos por citogenética convencional con los de la técnica FISH se observó que 2 de los 6 casos con cariotipo anormal (01 y 03) tenían alteraciones secundarias detectadas por FISH (Tabla n.º 2).
Los tres casos con hipodiploidías (01,12 y 15) no tenían aneuploidías del cromosoma 17 cuando se evaluaron por FISH. Sin embargo, en el caso 01 se detectó por FISH la deleción del gen TP53 (Tabla n.º 2).
De los casos que tenían translocaciones (02, 03, 14), dos fueron normales para el número de copias del cromosoma 17 y del gen TP53 por FISH (Tabla n.º 2); pero se obtuvo un resultado llamativo en el caso 03, que además de la t(9;22) identificada por citogenética convencional, tenía monosomía del cromosoma 17 y deleción del gen TP53 detectadas por FISH (Tabla n.º 2). Es decir, que en este caso se observaron tres alteraciones cromosómicas diferentes.
Por otra parte, en las dos muestras en las que no se obtuvieron metafases para el estudio cromosómico (casos 05 y 08), la técnica FISH en núcleos interfásicos reveló en el caso 05 una monosomía del cromosoma 17, con un número normal de copias para el gen TP53; mientras que el caso 08 fue normal tanto para el número de copias del cromosoma 17 como para el gen TP53 (Tabla n.º 2).
En los casos 04 y 06 en los que solo se hizo estudio por FISH para evaluar la presencia del cromosoma Filadelfia y cuyos resultados fueron negativos (Ph1), se encontró en el análisis por FISH que el caso 04 tenía una tetrasomía del cromosoma 17 y deleción del gen TP53; el caso 06 fue diferente pues solo presentó la deleción del gen TP53 (Tabla n.º 2).
De los resultados obtenidos se concluyó que solamente 3 (20%) de las 15 muestras analizadas por citogenética convencional y por FISH (casos 07, 09 y 10) eran normales en todos los aspectos: cariotipo, número de copias del cromosoma 17 y número de copias del gen TP53.
DISCUSIÓN
En este estudio se utilizó la técnica FISH–bicolor para evaluar el número de copias del cromosoma 17 y la deleción en el locus 17p13.1 del gen TP53 en 15 pacientes con diversas neoplasias hematológicas. El análisis cromosómico por citogenética convencional en 11 casos mostró que 6 (54,5%) tenían un cariotipo anormal. Este porcentaje está dentro del rango informado en la literatura (30 a 65%) para los diferentes tipos de neoplasias hematológicas,8,9,15,16 y corrobora que las alteraciones cromosómicas son frecuentes en las mismas. De los estudios citogenéticos se concluye que las neoplasias hematológicas presentan un alto grado de inestabilidad cromosómica.
Otros autores también han descrito las alteraciones cromosómicas encontradas en este estudio (translocaciones y mosaicismos con clones hipodiploides).1,8,15 Los pacientes con las translocaciones recíprocas t(9;22) (casos 02 y 03) tenían diagnóstico citomorfológico de leucemia mieloide crónica (LMC). En este tipo de leucemia dicha translocación, o cromosoma Filadelfia, es un marcador cromosómico presente en el 95% de los casos;1 su detección tiene gran importancia en el diagnóstico, pronóstico y seguimiento de los pacientes con LMC.4 De otro lado, la translocación t(8;21) observada en el caso 14, que tenía diagnóstico de leucemia mieloide aguda, es una alteración que se encuentra entre 5 y 12% de los casos de este tipo de leucemia.1 Por lo tanto, las translocaciones representan alteraciones cromosómicas estructurales frecuentes en las leucemias y que intervienen de manera crítica en el desarrollo de estas neoplasias.
En cuanto a los pacientes que tenían un mosaicismo con clones hipodiploides, cabe mencionar que esta condición y especialmente la hipodiploidía (menos de 46 cromosomas) es muy común en los diferentes tipos de leucemias, y que en muchos casos se la considera como un marcador cromosómico importante para el pronóstico de la enfermedad.9,16,17
Por otra parte, en este trabajo no se observaron otros tipos de aneuploidías como monosomía o trisomía de los cromosomas 7, 8 y 12, que también son muy frecuentes en las neoplasias hematológicas.8,15 Con respecto a los resultados de la técnica FISH, en este estudio se encontró una frecuencia de aneuploidía del cromosoma 17 del 26,7% (4/15); el tipo de alteración más frecuente fue la monosomía (tres casos) y hubo un caso de tetrasomía. Estos resultados concuerdan con los informados en otros estudios y están dentro del rango descrito de porcentajes de alteraciones.11,18–20 Estos hallazgos confirman que la aneuploidía del cromosoma 17 no es muy frecuente en las neoplasias hematológicas, a diferencia de las aneuploidías de los cromosomas 5,7, 8 y 12 que están entre las más frecuentes,11,14–19 las cuales se adquieren durante la carcinogénesis hematológica. Sin embargo, a pesar del bajo porcentaje de la monosomía del cromosoma 17, debe tenerse en cuenta que en este cromosoma se localizan diversos tipos de genes, entre ellos protooncogenes, genes supresores de tumores y genes de reparación (ERBB2, HER2/neu, BRCA1, TOPO II, TP53), que son esenciales para la estabilidad genómica.12,13 Por lo tanto, la pérdida o ganancia en el número de copias del cromosoma 17 afecta la expresión de estos genes, lo que podría inducir el mecanismo de inestabilidad genética.
Por otra parte, la deleción del gen TP53 se encontró en 5 pacientes (33,3%). En las neoplasias hematológicas, a semejanza de lo observado para la aneuploidía del cromosoma 17, se encontró una baja incidencia de deleción en la región 17p13.1 del gen TP53, con un rango entre 8 y 30%.14,18,19,21,22 Sin embargo, la mayoría de los estudios han hallado porcentajes menores del 20%. Thornton y colaboradores,23 en un estudio realizado con FISH en 115 muestras de pacientes con leucemia linfocítica crónica encontraron una frecuencia del 12% de deleción del gen TP53; en otro estudio similar al anterior, Sindelárová y colaboradores11 informaron una frecuencia del 16% de esta deleción en 206 casos con este mismo tipo de leucemia. El porcentaje de deleción del gen TP53 hallado en este trabajo es más alto que el informado en estos dos estudios. Tal diferencia podría explicarse por el menor tamaño muestral de nuestro estudio. Lo anterior permitiría concluir que la deleción del gen TP53 no es muy frecuente en las neoplasias hematológicas, a diferencia de los tumores sólidos en los que generalmente se presenta un alto porcentaje de esta deleción. Por otra parte, varios estudios informan que las deleciones más comunes en las neoplasias hematológicas son la 11q22 (ATM), 13q14 (RB), 7q–, 6q21 y 5q.7,11,18,22,24,25
En general podría considerarse que la deleción del gen TP53 constituye una alteración grave que afecta la estabilidad del genoma, por su importancia en el control del ciclo celular y en la reparación. Por lo tanto, la deleción, la inactivación por mutación o la no expresión de este gen promueven la aparición de diversas alteraciones genéticas en células neoplásicas, tal como se ha evidenciado en los tumores hematológicos.8,14,23
Adicionalmente, estudios epidemiológicos de neoplasias hematológicas indican que la deleción del gen TP53 se relaciona con un mal pronóstico de la enfermedad, una menor tasa de supervivencia y resistencia a ciertas drogas antineoplásicas (por ejemplo, análogos de la purina y agentes alquilantes).14.22,26–28 En resumen, las alteraciones en el gen TP53 tienen un papel importante en el comienzo y el avance del cáncer.
La técnica FISH fue informativa en los casos en que no se obtuvieron metafases para el estudio citogenético. El caso 05 presentó una monosomía del cromosoma 17, mientras que el caso 08 fue normal tanto para el número de copias del cromosoma 17, como para el gen TP53. Estos resultados, y otros discutidos más adelante, demuestran la importancia de la técnica FISH en núcleos interfásicos, especialmente cuando los cultivos celulares no son exitosos.29 Por tal razón, la técnica FISH se ha convertido en la última década en el 'estándar de oro' molecular para el diagnóstico citogenético de neoplasias hematológicas, y también para la investigación básica en cáncer.
Comparando los resultados obtenidos con la citogenética convencional y la técnica FISH, se encontraron hallazgos importantes. Los casos 11 y 13 tenían un cariotipo normal, pero el análisis por FISH detectó una deleción del gen TP53 (caso 11) y una monosomía del cromosoma 17 (caso 13). En este mismo sentido, los casos 01 y 03 presentaban un cariotipo anormal y por FISH se detectaron alteraciones cromosómicas adicionales. Se resalta el caso 03 porque fue el único en que se detectaron tres tipos de alteraciones genéticas. Una situación similar a la de los casos anteriores se observó en los casos 04 y 06, que tenían resultados negativos por FISH para la presencia del cromosoma Filadelfia y en los cuales se detectaron una tetrasomía del cromosoma 17 acompañada de deleción del gen TP53 (caso 04) y deleción del gen TP53 (caso 06). Estos resultados muestran que la técnica FISH, por sus altas sensibilidad y especificidad, tiene grandes ventajas sobre la citogenética convencional, puesto que permitió identificar alteraciones cromosómicas que no habían sido detectadas por la técnica convencional, lo que confirma su importancia en el diagnóstico citogenético molecular de estas neoplasias.3,5,18,30 Sin embargo, debe mencionarse que tampoco la técnica FISH–bicolor analiza la dotación cromosómica completa de la célula, puesto que solo identifica un determinado cromosoma o un tipo de alteración cromosómica estructural. Para resolver estas limitaciones existen variantes novedosas de la técnica FISH, como el SKY–FISH, M–FISH o CGH–arrays, que permiten evaluar muy detalladamente todos los cromosomas, y por lo tanto hacer un diagnóstico más completo y preciso en el análisis cromosómico de muestras tumorales.31
En la mayoría de las muestras analizadas con la técnica FISH se detectaron clones con núcleos heterogéneos (Tabla n.º 1), es decir, muestras que contenían subpoblaciones de núcleos monosómicos, disómicos, trisómicos y con menor frecuencia tetrasómicos. Lo anterior se explica por la heterogeneidad genética intratumoral, que es un mecanismo biológico común en el cáncer, básicamente originado por la inestabilidad genómica.32 La heterogeneidad genética y su expansión clonal se relacionan con el fenotipo tumoral y con la progresión, metástasis y resistencia a ciertas drogas antineoplásicas.32,33 En este trabajo, con la detección de subpoblaciones de núcleos con diferente dotación genética, se demostró la presencia de heterogeneidad en las muestras analizadas mediante la técnica FISH. Por tal motivo, se considera que esta técnica es muy eficiente en la detección de estos clones heterogéneos debido a la alta especificidad de las sondas.34 Además, tiene la gran ventaja de que con ella se puede analizar un mayor número de núcleos interfásicos por muestra (más de 100), comparado con el bajo número de metafases que se obtiene en las muestras de neoplasias hematológicas.
Finalmente, con este trabajo se concluye que las aneuploidías del cromosoma 17 y la deleción del gen TP53 en neoplasias hematológicas no constituyen alteraciones citogenéticas recurrentes. Otras alteraciones genéticas, que afectan a diferentes cromosomas y a múltiples genes, están implicadas directamente en el desarrollo de estas neoplasias hematológicas. Se requieren otros estudios para definir el valor pronóstico de las alteraciones del gen TP53 en los pacientes con estas neoplasias.
AGRADECIMIENTOS
Este trabajo fue financiado por el proyecto de investigación CPT–0118.
REFERENCIAS BIBLIOGRÁFICAS
1. Look AT. Genes altered by chromosomal translocations in leukemia and lymphomas. In: Vogeltein B, Kinzler KW, eds. The genetic basis of human genetic, 2ª ed. New York: McGraw–Hill; 2002. p. 57–92. [ Links ]
2. Croce CM. Oncogenes and cancer. N Engl J Med 2008; 358: 502–511. [ Links ]
3. Kolialexi A, Tsangaris GT, Kitsiou S, Kanavakis E, Mavrou A. Impact of cytogenetic and molecular cytogenetic studies on hematologic malignancies. Anticancer Res 2005; 25: 2979–2983. [ Links ]
4. Mauro MJ, Deininger MW. Chronic myeloid leukemia in 2006: a perspective. Haematologica 2006; 91: 152–158. [ Links ]
5. Kearney L. The impact of the new fish technologies on the cytogenetics of haematological malignancies. Br J Haematol 1999; 104: 648–658. [ Links ]
6. Levsky JM, Singer RH. Fluorescence in situ hybridization: past, present and future. J Cell Sci 2003; 116: 2833–2837. [ Links ]
7. Sreekantaiah C. FISH panels for hematologic malignancies. Cytogenet Genome Res 2007; 118: 284–296. [ Links ]
8. Dewald G, Ketterling R, Wyatt A, Stupca P. Cytogenetic studies in neoplastic hematologic disorders. En: McClatchey KD, ed. Clinical Laboratory Medicine, 2ª ed. Philadelphia: Lippincott Williams and Wilkins; 2005. p. 3–30. [ Links ]
9. Vásquez G, Ramírez GC, Cristancho CM, Durango NE, Ramírez JL. Experiencia diagnóstica en pacientes con diferentes tipos de leucemias, mediante citogenética convencional, 1998–2006. Iatreia 2006; 19: S10–S11. [ Links ]
10. Avet–Loiseau H, Li JY, Godon C, Morineau N, Daviet A, Harousseau JL, et al. P53 deletion is not a frequent event in multiple myeloma. Br J Haematol 1999; 106: 717–719. [ Links ]
11. Sindelárová I, Michalová K, Zemanová Z, Ransdorfová S, Brezinová J, Peková S, et al. Incidence of chromosomal anomalies detected with FISH and their clinical correlations in B–chronic lymphocytic leukemia. Cancer Genet Cytogenet 2005; 160: 27–34. [ Links ]
12. Strano S, Dell'Orso S, Di Agostino S, Fontemaggi G, Sacchi A, Blandino G. Mutant p53: an oncogenic transcription factor. Oncogene 2007; 26: 2212–2219. [ Links ]
13. Herrera JC, Vásquez G, Ramírez JL, Muñetón CM. Papel del gen TP53 en la oncogénesis. Salud UIS 2004; 26: 88–99. [ Links ]
14. Shaw GR, Kronberger DL. TP53 deletions but not trisomy 12 are adverse in B–cell lymphoproliferative disorders. Cancer Genet Cytogenet 2000; 119: 146–154. [ Links ]
15. Amiel A, Leopold L, Gronich N, Yukla M, Fejgin MD, Lishner M. The influence of different chromosomal aberrations on molecular cytogenetic parameters in chronic lymphocytic leukemia. Cancer Genet Cytogenet 2006; 167: 145–149. [ Links ]
16. Ripollés l, Ortega M, Ortuño F, González A, Losada J, Ojanguren J, et al. Genetic abnormalities and clinical outcome in chronic lymphocytic leucemia. Cancer Genet Cytogenet 2006; 171: 57–64. [ Links ]
17. Lessard M, Hélias C, Struski S, Perrusson N, Uettwiller F, Mozziconacci MJ. Fluorescence in situ hybridization analysis of 110 hematopoietic disorders with chromosome 5 abnormalities: do de novo and therapyrelated myelodysplastic syndrome–acute myeloid leukemia actually differ? Cancer Genet Cytogenet 2007; 176: 1–21. [ Links ]
18. Reddy KS. Chronic lymphocytic leukaemia profiled for prognosis using a fluorescence in situ hybridization panel. Br J Haematol 2006; 132: 705–722. [ Links ]
19. Sánchez J, Aventín A. Detection of chromosomal abnormalities in chronic lymphocytic leukemia increased by interphase fluorescence in situ hybridization in tetradecanoylphorbol acetatestimulated peripheral blood cells. Cancer Genet Cytogenet 2007; 175: 57–60. [ Links ]
20. Zhang L, Parkhurst JB, Cern WF, Scout KV, Niccum D, Mulvihill JJ, et al. Chromosomal changes detected by fluorescence in situ hybridization in patients with acute lymphoblastic leukemia. Chin Med J (Engl) 2003; 116: 1298–1303. [ Links ]
21. Fink SR, Smoley SA, Stockero KJ, Paternoster SF, Thorland EC, Van Dyke DL. Loss of TP53 is due to rearrangements involving chromosome region 17p10 approximately p12 in chronic lymphocytic leukemia. Cancer Genet Cytogenet 2006; 167: 177–181. [ Links ]
22. Xu W, Li JY, Wu YJ, Yu H, Shen QD, Li L, et al. Prognostic significance of ATM and TP53 deletions in Chinese patients with chronic lymphocytic leukemia. Leuk Res 2008; 32: 1071–1077. [ Links ]
23. Thornton P, Gruzka–Westwood AM, Hamoudi RA, Atkinson S, Kacsmarek P, Morilla ML, et al. Characterisation of TP53 in chronic lymphocytic leukaemia. Hematol J 2004; 5: 47–54. [ Links ]
24. Gozzetti A, Crupi R, Tozzuoli D, Raspadori D, Forconi F, Lauria F. Molecular cytogenetic analysis of B–CLL patients with aggressive disease. Hematology 2004; 9: 383–385. [ Links ]
25. Wiktor A, Van Dyke DL. Combined cytogenetic testing and fluorescence in situ hybridization analysis in the study of chronic lymphocytic leukemia and multiple myeloma. Cancer Genet Cytogenet 2004; 153: 73–76. [ Links ]
26. Peller S, Rotter V. TP53 in hematological cancer: low incidence of mutations with significant clinical relevance. Hum Mutat 2003; 21: 277–284. [ Links ]
27. Nahi H, Lehmann S, Bengtzen S, Jansson M, Möllgård L, Paul C, et al.Chromosomal aberrations in 17p predict in vitro drug resistance and short overall survival in acute myeloid leukemia. Leuk Lymphoma 2008; 49: 508–516. [ Links ]
28. Christiansen DH, Andersen MK, Pedersen–Bjergaard J. Mutations with loss of heterozygosity of p53 are common in therapy–related myelodysplasia and acute myeloid leukemia after exposure to alkylating agents and significantly associated with deletion or loss of 5q, a complex karyotype, and a poor prognosis. Clin Oncol 2001; 19: 1405–1413. [ Links ]
29. Cuneo A, Bigoni R, Roberti MG, Bardi A, Balsamo R, Piva N, et al. Detection of numerical aberrations in hematologic neoplasias by fluorescence in situ hybridization. Haematologica 1997; 82: 85–90. [ Links ]
30. Glassman AB, Hayes KJ. The value of fluorescence in situ hybridization in the diagnosis and prognosis of chronic lymphocytic leukemia. Cancer Genet Cytogenet 2005; 158: 88–91 [ Links ]
31. Kearney L, Horsley SW. Molecular cytogenetics in haematological malignancy: current technology and future prospects. Chromosoma 2005; 114: 286–294. [ Links ]
32. Lengauer C, Kinzler KW, Vogelstein B. Genetic instabilities in human cancers. Nature 1998; 396: 643–649. [ Links ]
33. Duesberg P, Rausch C, Rasnick D, Hehlmann R. Genetic instability of cancer cells is proportional to their degree of aneuploidy. Proc Natl Acad Sci USA 1998; 95: 13692–13697. [ Links ]
34. Galteland E, Holte H, Stokke T. –MYC, RB–1, TP53, and centromere 8 and 17 copy number in B–cell non–Hodgkin's lymphomas assessed by dual–color fluorescence in situ hybridization. Cytometry 1999; 38: 53–60. [ Links ]
Recibido: mayo 23 de 2008
Aceptado: septiembre 05 de 2008